Existuje široká škála linuxových bioinformatických nástrojů, které jsou v této oblasti již dlouhou dobu široce používány. Bioinformatika byla charakterizována mnoha způsoby; často se však definuje jako kombinace matematiky, počítání a statistiky pro analýzu biologických informací. Hlavním cílem bioinformatického nástroje je vyvinout účinný algoritmus, aby bylo možné odpovídajícím způsobem měřit podobnosti sekvencí.
Nejlepší bioinformatické nástroje pro Linux
Tento článek byl napsán se zaměřením na bioinformatické nástroje, které jsou dostupné na platformě Linux. Všechny účinné nástroje byly podrobně probrány a přezkoumány. Kromě toho v tomto článku najdete základní funkce, vlastnosti a odkazy ke stažení. Pojďme si to tedy projít.
1. geWorkbench
geWorkbench lze vypracovat pomocí genomového workbench je bioinformatický nástroj založený na java, který funguje pro integrovanou genomiku. Jeho komponenty architektury usnadňují specificky vyvinuté plug-iny, které by bylo možné konfigurovat do komplikovaných bioinformatických aplikací. V současné době je k dispozici více než sedmdesát zásuvných modulů pro podporu, vizualizaci a analýzu sekvenčních dat.
Funkce geWorkbench
- Je součástí mnoha nástrojů pro výpočetní analýzu, jmenovitě t-test, samoorganizující se mapy a hierarchické shlukování atd.
- Je vybaven sítěmi molekulárních interakcí, strukturou proteinů a daty o proteinech.
- Nabízí genovou integraci a anotační cesty a shromažďuje data z vybraných zdrojů pro analýzu obohacení genové ontologie.
- V tomto nástroji se komponenty integrují s platformou správy vstupů a výstupů.
2. BioPerl
BioPerl je sbírka nástrojů Perl široce používaných na platformě Linux jako bioinformatický nástroj pro výpočetní molekulární biologii. Neustále se používá v oblastech bioinformatiky do souboru standardního stylu CPAN. Tento linuxový bioinformatický nástroj je dobře zdokumentován a volně dostupný v modulech Perl. Vzhledem k tomu, že jsou objektově orientované, jsou tyto moduly při plnění úkolu vzájemně závislé.
Funkce BioPerl
- Z místních a izolovaných databází má tento bioinformatický nástroj přístup k údajům o nukleotidové a peptidové sekvenci.
- Manipuluje s odlišnými sekvencemi spolu s transformací formy databáze a záznamu souboru.
- Funguje jako bioinformatický vyhledávač, kde hledá podobné sekvence, geny a další struktury na genomové DNA.
- Tím, že generuje a manipuluje se zarovnáním sekvencí, vytváří strojově čitelné sekvenční anotace.
3. UGENE
UGENE je bezplatný open source a sada integračních bioinformatických nástrojů pro Linux. Jeho běžné uživatelské rozhraní je integrováno s většinou používaných a dobře známých bioinformatických aplikací. S jeho nástroji je kompatibilní řada formátů biologických dat; tak lze data získávat ze vzdálených zdrojů. Tento bioinformatický nástroj využívá vícejádrové CPU a GPU k poskytování maximálního možného výkonu pro optimalizaci svých výpočetních aktivit.
Funkce UGENE
- Jeho uživatelské uživatelské rozhraní nabízí několik funkcí, například vizualizaci chromatogramu, editor vícenásobného zarovnání a vizuální a interaktivní genomy.
- Vydláždí cestu pro 3D zobrazení ve formátech PDB a MMDB spolu s podporou anaglyfického stereo režimu.
- Usnadňuje zobrazení fylogenetického stromu, vizualizaci bodového grafu a návrhář dotazů může vyhledávat složité vzory anotací.
- To může připravit cestu pro vlastní výpočetní pracovní postup pro návrháře pracovních postupů.
4. Biojava
Biojava je otevřený zdroj a je navržen výhradně pro projekt, aby poskytoval požadované nástroje Java pro zpracování biologických dat. Funguje pro širokou škálu datových sad, například analytické a statistické rutiny, analyzátory pro běžné formáty souborů. Navíc usnadňuje manipulaci se sekvencí a 3D strukturou. Tento bioinformatický nástroj pro Linux má za cíl urychlit rychlý vývoj aplikací pro biologické datové sady.
Funkce Biojavy
- Včetně souborů tříd a objektů je to balíček, který implementuje kód Java pro různé datové sady.
- Biojavu lze použít v různých projektech, jako jsou Dazzel, Bioclips, Bioweka a Genious, které se používají k různým účelům.
- Funguje pro analyzátory souborů spolu s podporou klientů DAS a serveru.
- Používá se k provádění sekvenční analýzy pro GUI a má přístup k databázím BioSQL a Ensembl.
5. Biopython
Pro biologické výpočty se používá bioinformatický nástroj Biophython vyvinutý mezinárodním týmem vývojářů a napsaný v programu python. Nabízí přístup k široké škále formátů bioinformatických souborů, jmenovitě BLAST, Clustalw, FASTA, Genbank, a umožňuje přístup k online službám, jako je NCBI a Expasy.

Funkce Biopythonu
- Shromažďuje se s moduly python, které pracují na vytvoření sekvence s interaktivní a integrovanou povahou.
- Tento bioinformatický nástroj může pracovat v různých sekvencích, například při translaci, transkripci a výpočtech hmotnosti.
- Tento nástroj je výhradně obohacený; struktura proteinu a formát sekvence jsou tak řízeny efektivně.
- Tento linuxový bioinformatický nástroj funguje pro zarovnání; lze tedy vytvořit standard pro vytváření a zpracování substitučních matic.
6. InterMine
InterMine je open-source bioinformatický nástroj pro Linux, který funguje jako datový sklad pro integraci a analýzu biologických dat. Jako software si jej uživatelé mohou nainstalovat do svého zařízení a zpřístupnit data na webové stránce. Má se za to, že jde o jednu z nejdynamičtějších datových tabulek, která může snadno proniknout do dat a usnadňuje způsob filtrování dat. Který další sloupec slouží k navigaci na stránku přehledu?
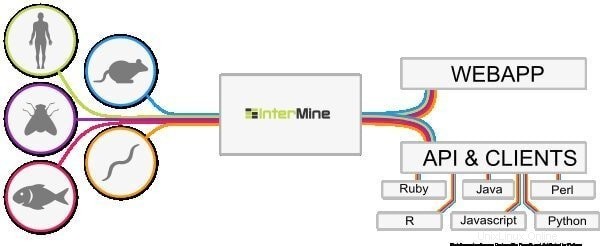
Funkce InterMine
- Funguje s jedním objektem, například genem, proteinem nebo vazebným místem, a více seznamy, jako je seznam genů nebo seznam proteinů.
- Může být provozován ve více jazycích; různé dotazy týkající se biometrických informací lze tedy vyhledávat v několika jazycích.
- V tomto softwaru jsou k dispozici čtyři vyhledávací nástroje:vyhledávání šablon, vyhledávání klíčových slov, nástroj pro tvorbu dotazů a vyhledávání regionů.
- Podporuje různé formáty, jako jsou Chado, GFF3, FASTA, GO a soubory asociace genů, UniProt XML, PSI XML, In Paranoid orthologs a Ensembl.
7. IGV
IGV, zpracovaný jako interaktivní prohlížeč genomiky, je považován za jeden z nejúčinnějších vizualizačních nástrojů, které mohou snadno přistupovat k rozsáhlé a interaktivní databázi genomiky. Může nabídnout širokou škálu datových typů s genomickou anotací spolu s daty založenými na poli a sekvencí nové generace. Stejně jako Mapy Google dokáže procházet sadou dat a hladce přibližovat a posouvat po celém genomu.
Funkce IGV
- Nabízí flexibilní integraci širokého rozsahu souborů genomických dat, včetně čtení zarovnaných sekvencí, mutací, počtu kopií a tak dále.
- Urychluje to umožnit průzkum v reálném čase týkající se masivní podpůrné datové sady pomocí efektivních formátů souborů s více rozlišeními.
- Mezi stovkami a do určité míry až tisíci vzorků umožňuje simultánní vizualizaci různých typů dat.
- Umožňuje načítání datových sad z místních a vzdálených zdrojů, včetně cloudových datových zdrojů, za účelem sledování vlastních a veřejně dostupných genomických datových sad.
8. GROMACS
GROMACS je dynamický molekulární simulátor, který je součástí analytických a stavebních nástrojů. Je to balíček s všestranností a má v úmyslu pracovat na molekulární dynamice; například dokáže simulovat newtonovskou pohybovou rovnici stovek až tisíců částic. Byl naprogramován tak, aby fungoval na biochemických molekulách v dřívější fázi, konkrétně na proteinech a lipidech, spojených komplikovanými interakcemi.
Funkce GROMACS
- Tento linuxový informatický nástroj je uživatelsky přívětivý, obsahuje topologie a soubory parametrů a je napsán v čistém textu.
- Nebyl použit skriptovací jazyk; všechny programy jsou tedy provozovány pomocí jednoduchého rozhraní příkazového řádku pro vstupní a výstupní soubory.
- Pokud se něco pokazí, provede se mnoho chybových zpráv a kontrola konzistence.
- Všechny programy jsou usnadněny pomocí integrovaného grafického uživatelského rozhraní.
9. Taverna Workbench
Taverna Workbench je open-source nástroj, který je naprogramován k navrhování a provádění bioinformatických pracovních postupů vytvořených projektem myGrid. S tímto nástrojem lze integrovat řadu softwaru, včetně SOAP a webové služby REST. Spolupracuje s různými organizacemi, jako je Evropský bioinformatický institut, DNA Databanka Japonska, Národní centrum pro biotechnologické informace, SoapLab, BioMOBY a EMBOSS.
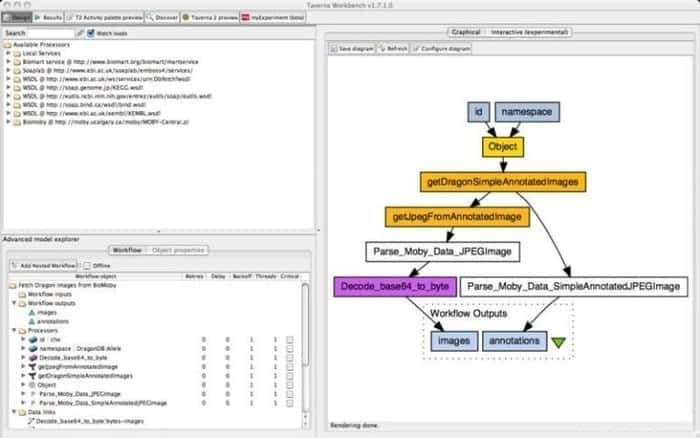
Funkce Taverna Workbench
- Je zcela navržen s grafickým pracovním postupem pro vyhledávání, vývoj a spouštění pracovních postupů.
- Byl navržen s plně grafickým pracovním postupem; navíc se pro návrh používají diskrétní záložky.
- Jsou uvedeny anotace pro popis pracovních postupů, služeb, vstupů a výstupů s vestavěnou nápovědou.
- Dříve použitý pracovní postup je uložen v tomto nástroji, i když může uložit vstupní pracovní postup použitý v souboru.
10. EMBOSSa
EMBOSS, který zahrnuje otevřenou softwarovou sadu European Molecular Biology. Jedná se o balík softwaru, který byl vyvinut pro potřeby komunity molekulární biologie. Tento linuxový bioinformatický nástroj lze použít pro různé účely. Například je funkční v různých formátech dat automaticky. Navíc může shromažďovat data postupně z webové stránky.
Funkce EMBOSS
- EMBOSS je součástí stovek aplikací, jmenovitě zarovnání sekvencí a rychlého vyhledávání v databázi pomocí vzorců sekvencí.
- Navíc má identifikaci proteinového motivu, včetně analýzy domény a analýzy vzoru nukleotidové sekvence.
- Jeho sada nástrojů byla navržena tak, aby odpovídala bioinformatické aplikaci a pracovnímu postupu.
- Byla naprogramována s dalšími knihovnami, aby zvládla také mnoho dalších relevantních problémů.
11. Clustal Omega
Clustal Omega pracuje na proteinu a RNA/DNA je program pro zarovnání více sekvencí určený pro obecné účely. Dokáže efektivně zpracovat miliony datových sad v rozumném čase; navíc vyrábí vysoce kvalitní MSA. V tomto linuxovém bioinformatickém nástroji existuje proces, kdy uživatel vyžaduje ponechat sekvenci souborů ve výchozím režimu. To se zarovná a seskupí, aby se vygeneroval vodící strom, a to nakonec umožní vytvořit sekvenci postupného zarovnání.
Funkce Clustal Omega
- Usnadňuje vzájemné zarovnání existujících zarovnání a co víc, zarovnání sekvence k zarovnání pro použití skrytého Markovova modelu.
- Existuje funkce, která se nazývá externí zarovnání profilu, která odkazuje na novou sekvenci homologní pro skrytý Markovův model.
- HMM se používají pro Clustal Omega pro seřizovací stroj převzatý z balíčku HHalign od Johannese Soedinga.
- Clustal Omega umožňuje tři typy sekvenčních vstupů:profil, zarovnání sekvence a HMM.
12. BLAST
Basic Local Alignment Search Tool nebo BLAST se používá pro nalezení podobnosti mezi biologickými sekvencemi. Dokáže najít relevantní shody mezi nukleotidovými a proteinovými sekvencemi a ukázat jejich statistickou důležitost. Sekvence dotazů jsou strukturovány s různými typy BLAST. A co víc, tento nástroj je z velké části kultivován prosperujícími neznámými geny u různých zvířat a umožňuje mapovat soubory dat založené na sekvencích pomocí kvalitativní analýzy.

Funkce BLAST
- Nukleotid-nukleotid megaBLAST nabízí vyhledávání a optimalizaci velmi podobných typů sekvencí.
- Navíc nukleotid-nukleotid BLASTN funguje trochu jiným způsobem, protože hledá sekvence vzdálenosti.
- A co víc, BLASTP provádí hledání vztahu protein-protein a porovnání a jeho vzorec se používá pro různé další výzkumy.
- TBLASTN se zaměřuje na dotazování nukleotidů na datovou sadu proteinů a dokáže překládat databázi za běhu.
13. Postýlka
Bioinformatický software Bedtool je švýcarský armádní nůž nástrojů používaných pro širokou škálu genomických analýz. Genomická aritmetika používá tento nástroj velmi široce, což znamená, že s ním dokáže najít teorii množin. Bedtools například usnadňují počítání, doplňování a míchání, protínání, slučování genomických intervalů z více souborů a generování určitého formátu genomu, jako je BAM, BED, GFF/GTF, VCF.
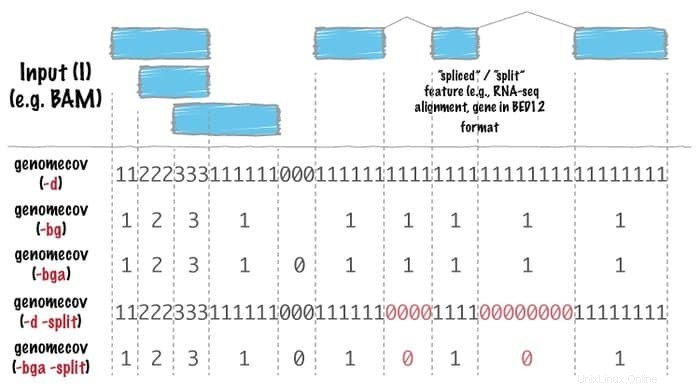
Funkce Bedtools
- V tomto linuxovém bioinformatickém nástroji je každý navržen tak, aby vykonával zvlášť jednoduchý úkol, např. protínal dva intervalové soubory.
- Komplikovaná a sofistikovaná analýza se provádí pomocí kombinace nástrojů.
- Tento nástroj byl vyvinut v laboratoři Quinlan University v Utahu skupinovým výzkumníkem.
- Vzhledem k tomu, že tento nástroj obsahuje mnoho možností, lze jej použít pro víceúčelové účely v oblasti bioinformatiky.
14. Bioklipsa
Bioinformatický nástroj Bioclipse Linux, který je definován s pracovním stolem pro vědy o živé přírodě, je open-source software založený na java. Funguje na vizuální platformě, která zahrnuje chemo a bioinformatiku Eclipse Rich Client Platform. Je vybaven architekturou pluginů. To znamená nejmodernější architekturu pluginů, funkčnost a vizuální rozhraní z Eclipse, jako je systém nápovědy, včetně aktualizací softwaru.
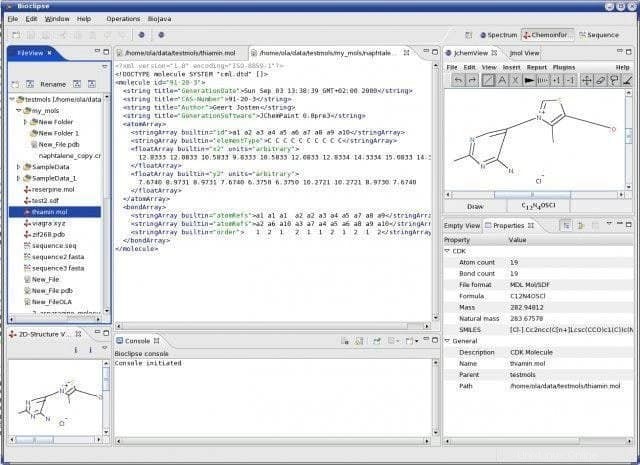
Funkce Bioclipse
- Biologické sekvence, konkrétně RNA, DNA a protein, jsou spravovány pomocí bioklipsu.
- Biojava také pomáhá při poskytování základních funkcí bioinformatiky; také grafické editory pro zarovnání sekvencí.
- Používá se pro farmakologii a objevování léků spolu s místem objevu metabolismu.
- Nakonec funguje na funkčnosti sémantického webu, procházení rozsáhlých sbírek sloučenin a úpravy chemických struktur.
15. Biovodiča
Bioinformatika široce používaná na platformě Linux je open source a bezplatný bioinformatický nástroj, který se koherentně používá v lékařské biologii pro vysoce výkonnou analýzu. Používá především statistické programování R; nicméně obsahuje také další programovací jazyk. Tento software je navržen se zaměřením na několik cílů; například si klade za cíl vytvořit společný vývoj a zajistit používání inovativního softwaru v obrovské míře.
Funkce Bioconductor
- Tento software dokáže analyzovat řadu dat, například pole oligonukleotidů, sekvenční analýzu, průtokový cytometr a může generovat robustní grafickou a statistickou databázi.
- Mít viněty a dokumenty v každém a binokulárním balíčku může poskytnout textový a úkolově orientovaný popis funkčnosti tohoto balíčku.
- Může generovat data v reálném čase týkající se přidružení microarray a dalších genomických dat spolu s biologickými metadaty.
- Navíc může analyzovat expresní geny, jako jsou LIMMA, cDNA Arrays, Affy Arrays, RankProd, SAM, R/maanova, Digital Gene Expression a tak dále.
16. AMFORA
AMPHORA, což je zkratka pro Automated Phylogenomic InfeRence Application, je open source nástroj pro pracovní toky bioinformatiky. Další verze AMPHORA, která se nazývá AMPHORA2, má bakteriální a 104 archaálních fylogenetických markerových genů. Ještě důležitější je, že funguje na vytváření informací mezi fylogenetickými a met genetickými datovými sadami.
Funkce AMPHORA
- Protože se jedná o jednotlivé geny, je AMPHORA2 nejvhodnější pro odvození taxonomického složení bakterií.
- Navíc také dokáže odvodit taxonomické složení archaálních společenstev z metagenomické sekvence brokovnic.
- Zpočátku se AMPHORA používala k analýze metagenomických dat Sargasového moře.
- Avšak v současnosti se AMPHORA2 stále více používá k analýze relevantních metagenomických dat v tomto ohledu.
17. Andurila
Anduril je bioinformatický software založený na komponentách s otevřeným zdrojovým kódem pro Linux, který slouží k vytvoření rámce pracovních postupů týkajících se analýzy vědeckých dat. Tento nástroj vyvinula Systems Biology Laboratory, University of Helsinki. Tento bioinformatický nástroj pro Linux je navržen tak, aby umožňoval efektivní, flexibilní a systematickou analýzu dat, zejména v oblasti biomedicínského výzkumu.
Funkce Abdurila
- Funguje v pracovním postupu, kde jsou vzájemně propojeny různé systémy zpracování; například; výstup procesu může fungovat jako vstup ostatních.
- Primární nástroj Anduril je napsán v jazyce Java, zatímco ostatní komponenty jsou napsány v různých aplikacích.
- V jeho různých krocích probíhají četné činnosti, jako např. vytváří data, generuje sestavy a také importuje data.
- Konfiguraci pracovního postupu lze provést pomocí jednoduchého otevřeného a výkonného skriptovacího jazyka, jmenovitě Andurilscript.
18. LabKey Server
LabKey Server je preferovanou volbou pro vědce používané v laboratořích k integraci výzkumu, analýzy a sdílení biomedicínských dat. V tomto nástroji je použito zabezpečené úložiště dat, které usnadňuje webové dotazy, hlášení a spolupráci v rámci široké řady databází. Spolu s danou základní platformou lze do této aplikace přidat mnoho dalších vědeckých nástrojů.
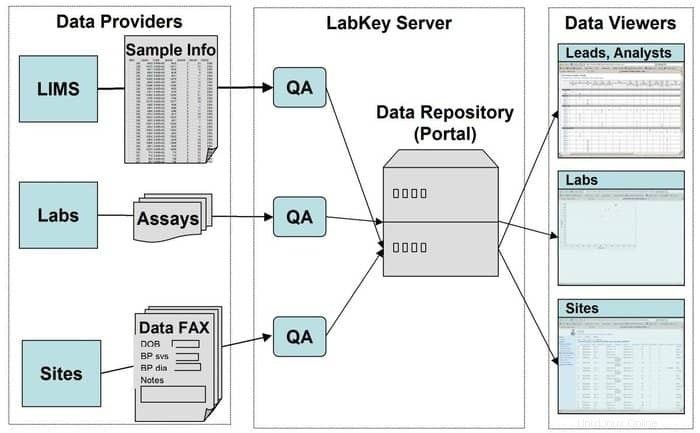
Funkce LabKey Server
- LabKey Server obsahuje všechny typy biomedicínských dat. Například průtoková cytometrie, mikročipy, hmotnostní spektrometrie, mikrodestičky, ELISpot, ELISA atd.
- V tomto nástroji provádí přizpůsobitelný kanál zpracování dat všechny relevantní činnosti.
- Je vybaven observačními studiemi, které podporují řízení longitudinálních rozsáhlých studií účastníků.
- Proteomika se používá ke zpracování dat hmotnostní spektrometrie s vysokou propustností pomocí specifického nástroje, jmenovitě X! Tandem.
19. Mothur
Mothur je open-source bioinformatický nástroj široce používaný v biomedicínské oblasti pro zpracování biologických dat. Jedná se o softwarový balík, který se často používá pro analýzu DNA z nekultivovaných mikrobů. Mothur je linuxový bioinformatický nástroj, který dokáže zpracovat data generovaná metodami sekvence DNA, včetně 454 pyrosekvenování.
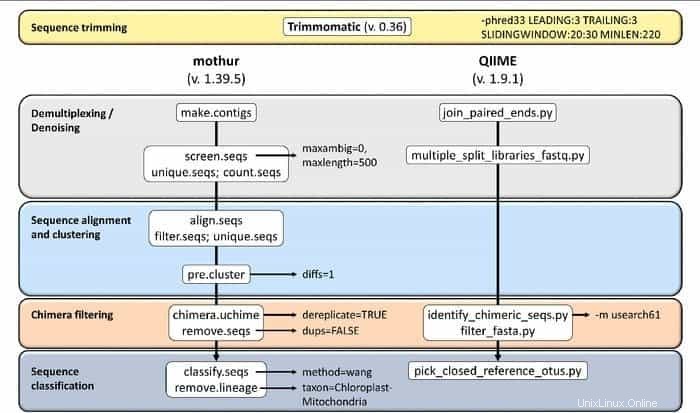
Funkce Mothur
- Jedná se o jediný balík softwaru, který je schopen zpracovat analýzu dat komunity a vytvořit sekvenci.
- Tento nástroj poskytuje rozsáhlou komunitní dokumentační podporu a další formy podpory.
- Předpokládá se, že Mothur je nejvýznamnější bioinformatický nástroj analyzující sekvence genu 16S rRNA.
- V tomto nástroji je k dispozici specializovaná komunita a výukové programy, které informují o tom, jak používat Sanger, PacBio, IonTorrent, 454 a Illumina (MiSeq/HiSeq).
20. VOTCA
VOTCA je zkratka pro Versatile Object-oriented Toolkit for Coarse-graining Applications, která je označena jako účinný bioinformatický nástroj s balíčkem pro hrubozrnné modelování, který analyzuje především molekulárně biologická data. Jeho cílem je vyvinout systematické hrubozrnné techniky spolu se simulací mikroskopického náboje pro transport neuspořádaných polovodičů.
Funkce VOTCA
- VOTCA obsahuje hlavně tři hlavní části:sadu nástrojů pro hrubé zrnění, sadu nástrojů Charge Transport a sadu nástrojů Excitation Transport Toolkit.
- Všechny tři základní funkce pocházejí z knihovny nástrojů VOTCA, která implementuje sdílené procedury.
- VOTCA používá hrubozrnné metody k získání nejlepších výsledků z relevantních činností.
- Tento software je vybaven sadou nástrojů pro přenos excitace, kde do značné míry podporuje balíčky orca DFT.
Poslední myšlenka
Abychom to celé shrnuli, stojí za zmínku, že všechny čtvrté zmíněné bioinformatické aplikace jsou v této oblasti široce využívány. Tyto linuxové bioinformatické nástroje se již dlouhou dobu používají v lékařské vědě, farmakologii, vynálezech léků a příslušné sféře. Nakonec vás žádáme, abyste nechali své dva haléře týkající se tohoto článku. A co víc, pokud zjistíte, že tento článek stojí za to, nezapomeňte jej lajkovat, sdílet a komentovat. Váš cenný komentář oceníme.